von kevin guevara Vor 4 Jahren
277
Organigrama arbol
von kevin guevara Vor 4 Jahren
277

Mehr dazu
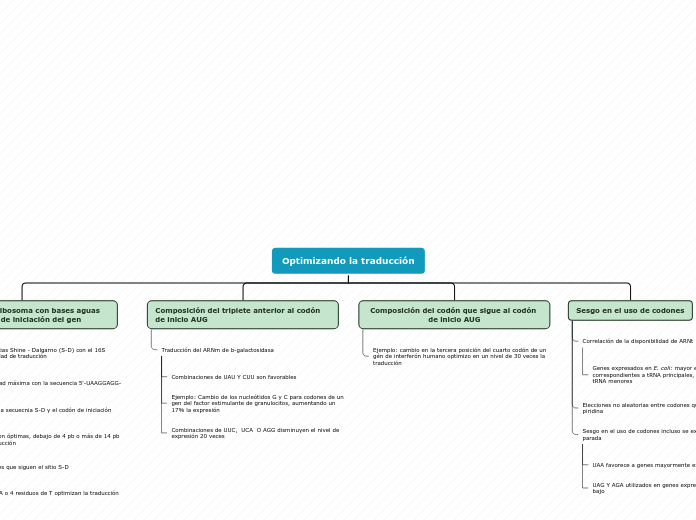
4 residuos de A o 4 residuos de T optimizan la traducción
Ocho bases son óptimas, debajo de 4 pb o más de 14 pb reduce la traducción