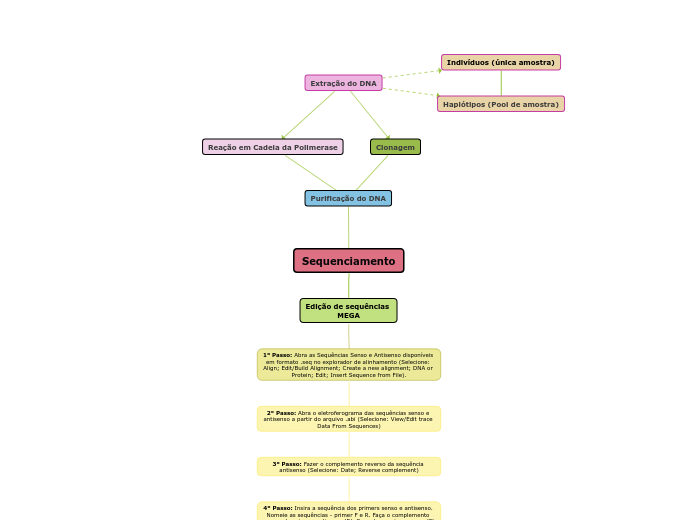
Sequenciamento
Edição de sequências
MEGA
1º Passo: Abra as Sequências Senso e Antisenso disponíveis em formato .seq no explorador de alinhamento (Selecione: Align; Edit/Build Alignment; Create a new alignment; DNA or Protein; Edit; Insert Sequence from File).
2º Passo: Abra o eletroferograma das sequências senso e antisenso a partir do arquivo .abi (Selecione: View/Edit trace Data From Sequences)
3º Passo: Fazer o complemento reverso da sequência antisenso (Selecione: Date; Reverse complement)
4º Passo: Insira a sequência dos primers senso e antisenso. Nomeie as sequências - primer F e R. Faça o complemento reverso do primers antisenso (R). Encontre o primer senso (F) no início da sequência antisenso e o primer antisenso (R) no final da sequência senso. Exclua os primers e as sequências alinhadas a eles (Selecione: o símbolo +; copie e cole as sequências em cada linha; Date; Reverse complement).
5º Passo: Faça o alinhamento das sequÊncias (Selecione: W; Align DNA; yes; Ok).
6º Passo: com os eletroferogramas abertos, faça o complemento reverso do eletroflerograma antisenso.
7º Passo: Verifique a região de baixa qualidade e exclua as sequências/bases dessa região.
8º Passo: Clique em uma das sequÊncias do explorador de alinhamento e gere uma fita consenso - copie e cole.
9º Passo: Analise pelos eletroferogramas as regiões das sequências que apresentem gaps, bases diferentes no alinhamento (IUPAC) e de forma conservacionista edite as sequências da fita consenso.
10º Passo: Exporte o arquivo no formato FASTA e salve-o.