Replicación de ADN
Eucariontes
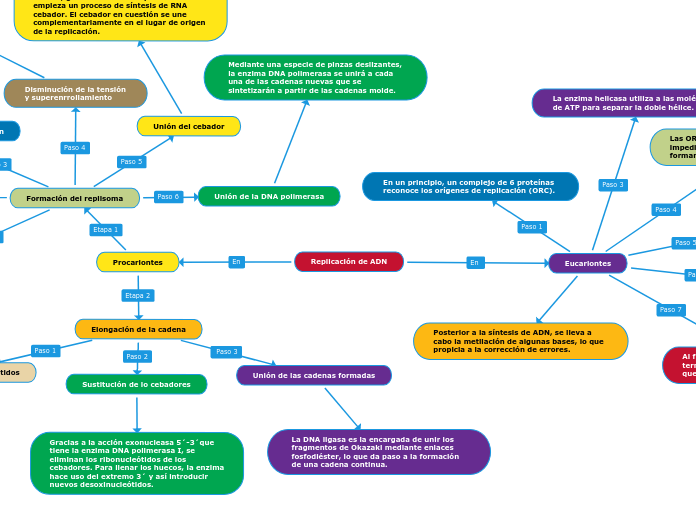
En un principio, un complejo de 6 proteínas reconoce los orígenes de replicación (ORC).
La enzima helicasa utiliza a las moléculas de ATP para separar la doble hélice.
Las ORC se unen a las dos cadenas para impedir que las hebras vuelvan a unirse, formando ojales de replicación.
A cada horquilla se le une una DNA polimerasa, comenzando así con el proceso de la síntesis de pequeños fragmentos de RNA con aproximadamente 20 nucleótidos.
Otra DNA polimerasa alarga la cadena en dirección 5´a 3´, es decir que, solo servirá para generar la cadena que utiliza como molde la dirección 3´a 5´; mientras que la cadena que utiliza como molde la dirección 5´a 3´, se formará mediante los fragmentos de Okazaki.
Al final, las dos horquillas que se acercaban terminan por unirse para formar una sola, quedando las dos cadenas entrelazadas.
Posterior a la síntesis de ADN, se lleva a cabo la metilación de algunas bases, lo que propicia a la corrección de errores.
Procariontes
Formación del replisoma
Identificación del sitio de replicación
El sitio de replicación de ADN es un lugar específico que depende directamente del tipo de microorganismo.
Apertura de la doble cadena
Las proteínas DnaA se unen a las moléculas de ATP de tal manera que, en conjunto hacen posible que se abra una agrupación de secuencias enriquecidas en AT. Con esto, se facilita la unión de la enzima helicasa que tiene como función abrir aún más la hélice.
Estabilización de la horquilla de replicación
Una horquilla de replicación es básicamente el resultado del paso anterior, es decir, es el origen de la abertura que va moviéndose a lo largo del ADN. Para su estabilización se le unen las SSBP.
Disminución de la tensión y superenrrollamiento
En esta etapa se genera un superenrrollamiento causado por una tensión de enrrollamiento por delante de la horquilla. Para disminuir este efecto, interviene la enzima DNA girasa. Su función es la de hacer cortes en las cadenas de ADN y catalizar movimientos locales para deshacer los giros y los nudos que se formaron el el superenrrollamiento.
Unión del cebador
A partir de que la doble hélice se encuentra abierta, gracias a la enzima primasa empieza un proceso de síntesis de RNA cebador. El cebador en cuestión se une complementariamente en el lugar de origen de la replicación.
Unión de la DNA polimerasa
Mediante una especie de pinzas deslizantes, la enzima DNA polimerasa se unirá a cada una de las cadenas nuevas que se sintetizarán a partir de las cadenas molde.
Elongación de la cadena
Adición de desoxirribonucleótidos
Para ambas cadenas, la DNA polimerasa III introduce nucleótidos a los RNA cebadores; la adenina siempre irá acompañada de una timina y la guanina de la citosina. Enseguida se formaran los puentes de hidrogeno entre las bases complementarias. Cabe aclarar que este proceso se da en la dirección 5´ a 3´.
Sustitución de lo cebadores
Gracias a la acción exonucleasa 5´-3´que tiene la enzima DNA polimerasa I, se eliminan los ribonucleótidos de los cebadores. Para llenar los huecos, la enzima hace uso del extremo 3´ y así introducir nuevos desoxinucleótidos.
Unión de las cadenas formadas
La DNA ligasa es la encargada de unir los fragmentos de Okazaki mediante enlaces fosfodiéster, lo que da paso a la formación de una cadena continua.