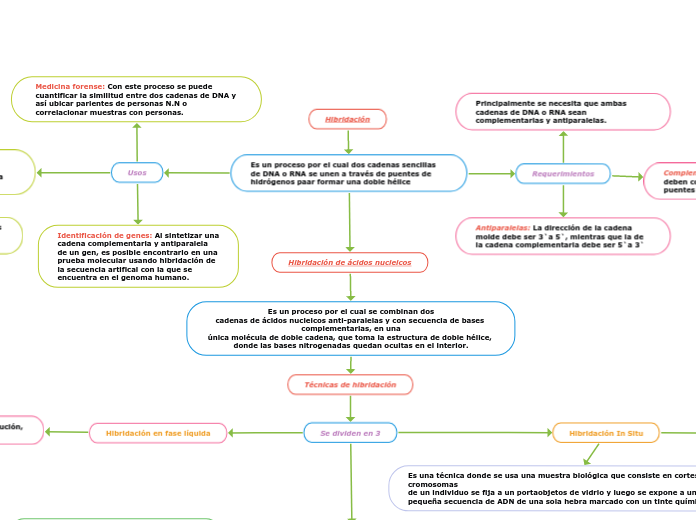
Hibridación de ácidos nucleicos
Es un proceso por el cual se combinan dos
cadenas de ácidos nucleicos anti-paralelas y con secuencia de bases complementarias, en una
única molécula de doble cadena, que toma la estructura de doble hélice, donde las bases nitrogenadas quedan ocultas en el interior.
Técnicas de hibridación
Se dividen en 3
Hibridación en fase líquida
Esta utiliza una muestra de DNA o RNA, en disolución,
además se realiza la hibridación con la sonda
Hibridación en soporte sólido
Permite procesar varias muestras de forma simultánea y facilita el control de la hibridación.
Son válidas para el análisis de ácidos nucleicos y el diagnóstico de enfermedades
moleculares con base genética.
Es el método empleado para identificar, de entre varias colonias bacterianas en placas de cultivo con agar, las que contienen un gen o fragmento de DNA de interés bajo la forma de plásmido recombinante
Se divide en
Dot - blot y Slot - blot
Es el método más simple y común, gracias a la posibilidad de emplearlo con muestras de ácido nucleico sin purificar.
La muestra se aplica, bien gota a gota (dot-blot) o en manchas alargadas (slot.blot)
sobre un "filtro" de nailon o nitrocelulosa, cuyas propiedades de adsorción fijan las moléculas de ácido nucleico y proteína.
Northern blot
Para uniones ADN-ADN
Se prepara ADN con enzima de restricción y los fragmentos se separan por tamaños,
mediante electroforesis en un gel.
Luego los fragmentos son desnaturalizados y se separan las 2 hebras componentes de
ADN en sus cadenas sencillas.
Posteriormente, el ADN inserto en el gel, es transferido a un filtro de nitrocelulosa, y
queda representado una réplica de la disposición de los fragmentos de ADN presentes
en el gel.
Finalmente el filtro se incuba, con la sonda; durante la incubación, la sonda se va
hibridando a las moléculas de ADN de cadena sencilla de secuencia complementaria
Aplicaciones
Se lo utiliza para analizar fragmentos de DNA generados por digestión con enzimas de restricción.
Southern blot
Para uniones ADN- ARN
Se utiliza para identificar las cadenas de ARN de secuencia semejante al ADN que se
usa como sonda.
Se utiliza para obtener información sobre el tamaño de ARN
Además para conocer qué genes están activos formando ARNm, en qué tejidos o tipos
celulares.
En este el ARN se extrae y se fracciona en tamaños variables, mediante electroforesis en gel de
poliacrilamida o agarosa, en condiciones que mantenga las cadenas de ARN,
desnaturalizado.
Aplicaciones
Se utiliza para detectar la presencia de moléculas específicas de ARN en una muestra
específica.
Proporciona información sobre moléculas que hibridan. Por ejemplo, su tamaño
molecular.
Se utiliza para examinar patrones de expresión génica en tejidos embrionarios y en
adultos.
Se utiliza también para caracterizar y cuantificar la
actividad transcripcional de un gen determinado en distintas células, tejidos u
organismos.
Se usan generalmente para caracterizar la identidad de genes específicos, para
localizar regiones codificantes o regiones reguladoras flanqueantes en secuencias
clonadas. Por ende, sirve para investigar la organización molecular de las secuencias genómicas.
Hibridación In Situ
Es una técnica donde se usa una muestra biológica que consiste en cortes de tejido, células o cromosomas
de un individuo se fija a un portaobjetos de vidrio y luego se expone a una “sonda”, que es una
pequeña secuencia de ADN de una sola hebra marcado con un tinte químico o fluorescente.
Las sondas están marcadas con colorantes fluorescentes
Aplicaciones
Se realiza sobre cortes de tejido, células intactas, núcleos aislados, etc.
Es usado para detectar virus patógenos
Para diagnosticar células cancerosas como consecuencia de cambios genéticos
Para estudiar cambios en la expresión génica, detectando secuencias en el RNA celular
La detección se realiza por autorradiografía y examen de la película fotográfica bajo el
microscopio, o por microscopia de fluorescencia, si se empleó este marcaje para la sonda.
Hibridación In Situ cromosómica
Se realiza sobre preparaciones de cromosomas metafásicos o prometafásicos, tratadas con
reactivos fijadores para preservar las características morfológicas del cromosoma.
Se trata la muestra con enzimas proteolíticas y se desnaturaliza el DNA mediante calor,
álcali o formamida para permitir su hibridación con la sonda
Elevado potencial diagnóstico en cromosomopatías, genes tumorales, virología, trasplantes.
Hibridación
Es un proceso por el cual dos cadenas sencillas de DNA o RNA se unen a través de puentes de hidrógenos paar formar una doble hélice
Usos
PCR: En el proceso de reacción de cadena con polimerasa, en la primera etapa, se hace una hibridación del primer que utilizamos con la cadena de DNA que se usa
Así la localización de la secuencia de nucleótidos que se va multiplicar exponencialmente es específica.
Medicina forense: Con este proceso se puede cuantificar la similitud entre dos cadenas de DNA y así ubicar parientes de personas N.N o correlacionar muestras con personas.
Identificación de genes: Al sintetizar una cadena complementaria y antiparalela de un gen, es posible encontrarlo en una prueba molecular usando hibridación de la secuencia artifical con la que se encuentra en el genoma humano.
Requerimientos
Principalmente se necesita que ambas cadenas de DNA o RNA sean complementarias y antiparalelas.
Complementarias: Sus bases nitrogenadas deben complementarse de forma A-T (2 puentes de H) y G-C (3 puentes de H)
Antiparalelas: La dirección de la cadena molde debe ser 3`a 5`, mientras que la de la cadena complementaria debe ser 5`a 3`
Bibliografías
Karp, G. (2014). Biología celular y molecular: Conceptos y experimentos (7a ed. --.).
Biología. Curtis H., Barnes S., Schnek A. y Massarini A. (2008) 7a Edición. Editorial Médica Panamericana.
Salazar Montes, A. M., Sandoval Rodríguez, A. S., & Armendáriz Borunda, J. S. (2013).
Biología molecular: Fundamentos y aplicaciones en las ciencias de la salud (1a. ed.--.). México D.F.: McGraw Hill.