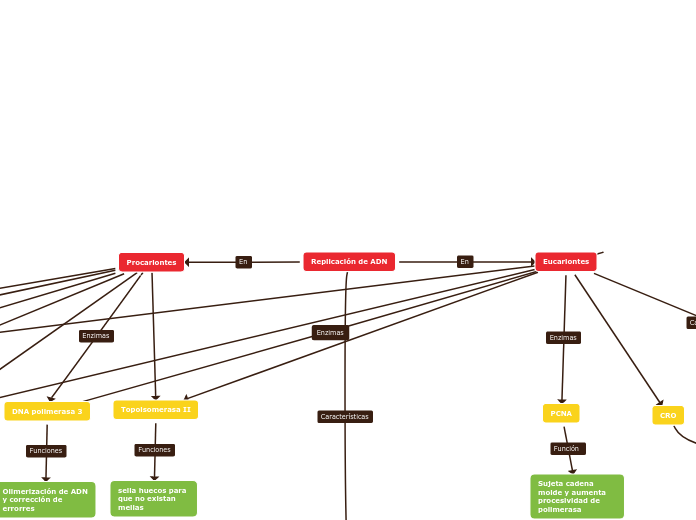
Replicación de ADN
semiconservatida, bidireccional, síntesis de cadenas discontinuas y continuas
Inicio
Para reconocer la secuncia 245pb se necesitan una serie de proteínas
Dam metilasa, Dna A, HU, Dna C
Elongación
Se extiende el cebador (adición de dexirribonucleótidos)catalizando enlaces fosfodiéster. se sintetizan las 2 cadenas
Helicasa, Proteína ssb, Topoisomerasa, Primasa, DNA polimerasa 3-1, Ligasa
Terminación
Termina por TER/TUR (especie de topes)
Eucariontes
PCNA
Sujeta cadena molde y aumenta procesividad de polimerasa
cromosomas lineales, varios sitios de origen (SRA) y 2 orquillas en cada sitio de origen
CRO
Procariontes
DNA polimerasa 3
Olimerización de ADN y corrección de errorres
2 nucleos de polimerizaación, 2 abrazaderas para sujetar molde, sub unidad epsilon (correciones), sub unidad teta (estabiliza unión), sub unidad Tau 8une a los nucleos)
Dam metilassa
Metila A en las secuencias de 13pb en tandem para que las enzimas la reconozcan e inicie la replicación
Primasa DNA G
Sintetiza cebador de polimerasa RNA y es dependiente de DNA
Dominio de unión, dominio de RNA y dominio de zinc
Helicasa
Separa la doble hélice del DNA para generar cadenas sencillas eliminando puentes de hidrogeno
Exomérica
Proteína ssb
estabiliza cadena sencilla para evitar la renaturalización
DNA polimerasa 1
polimeriza ADN, elimina cadenas y cataliza la formación de enlaces fosfodiéster.
ESTRUCTURA
Fragmento pequeño y klenow
Nucleasa
Exonucleasa
corte externo de DNA: 3´5´ - 5´3'
Endonucleasa
Cortes internos de DNA
Topoisomerasa II
sella huecos para que no existan mellas
Dos sub unidades: Gyr A- Gyr B