Base de datos biologicas

GENBANK
GENBANK: BASE DE DATOS DE LAS SECUENCIAS DE NUCLEOTIDOS Y PROTEÍNAS MÁS CONOCIDAS
GenBank es una base de datos compuesta por la mayoría secuencias públicas conocidas de ADN y proteínas (Benson et al., 2009). Además de almacenar estas secuencias, GenBank contiene anotaciones bibliográficas y biológicas
Cantidad de datos de secuencia
GenBank actualmente contiene alrededor de 100 mil millones de nucleótidos de 100 millones de secuencias.La división WGS consiste en secuencias generadas por esfuerzos de secuenciación de alto rendimiento. Desde 2002, las secuencias de WGS han estado disponibles en NCBI, pero no se consideran parte de los lanzamientos de GenBank. El número de pares de bases de ADN incluidos entre las secuencias de WGS (136 mil millones de pares de bases en la versión 168, octubre de 2008) es mayor que el tamaño de GenBank.
Organismos en GenBank
Más de 260,000 especies diferentes están representadas en GenBank, con más de 1000 nuevas especies agregadas por mes
TAXONOMÍA REPRESENTADA EN GENBANK
BCT Bacteriasa
ENV Muestras ambientales
INV Invertebrados
MAM Otros mamíferos
PHG Bacteriófagos
PLN Plantas
PRI Primates
ROD Roedores
SYN Sintético
GSS Genome survey sequences
Tipos de datos en GenBank
Bases de datos de ADN de GenBank que contiene datos de globina beta
no redundante (nr)
dbGSS
dbHTGS
dbSTS
Bases de datos de ADN de GenBank, derivado de ARN, que contiene datos de globina beta.
Gen entrez
dbEST
UniGene
Expresión génica Ómnibus
Bases de datos de proteínas que contiene datos de globina beta
Proteina Entrez
no redundante (nr)
UniProt
Bases de datos de ADN genómico
• La globina beta es parte de un cromosoma. En el caso de la RBP humana, veremos que su gen está situado en el cromosoma 11
• La globina beta puede ser parte de un gran fragmento de ADN, como un cósmido, un cromosoma artificial bacteriano (BAC) o un cromosoma artificial de levadura (YAC) que puede contener varios genes.
• La globina beta está presente en las bases de datos como un gen. El gen es la unidad funcional de la herencia y es una secuencia de ADN que típicamente consiste en regiones reguladoras, exones codificadores de proteínas e intrones. A menudo, los genes humanos son de 10 a 100 kb de tamaño.
Bases de Datos de ADNc Correspondientes a Genes Expresados
La globina beta se representa en las bases de datos como una etiqueta de secuencia expresada (EST), es decir, una secuencia de ADNc derivada de una biblioteca de ADNc particular. Si uno obtiene un tejido como el hígado, purifica el ARN y luego convierte el ARN en una forma más estable de ADNc, es probable que algunos de los clones de ADNc contenidos en ese ADNc codifiquen beta globina.
Etiquetas de secuencia expresada (ESTs)
La base de datos de etiquetas de secuencia expresada (dbEST) es una división de GenBank que contiene datos de secuencia y otra información sobre secuencias de cDNA de "un solo paso" de varios organismos
ESTs y UniGene
El objetivo del proyecto UniGene (gen único) es crear agrupaciones orientadas a genes mediante la partición automática de los EST en conjuntos no redundantes.
Sitios etiquetados de secuencia (STS)
El dbSTS es un sitio NCBI que contiene STS, que son secuencias de puntos de referencia genómicas cortas para las cuales se dispone de datos de secuencias de ADN y datos de mapeo
Secuencia genómica de alto rendimiento (HTGS)
La división HTGS se creó para que los datos de secuencias genómicas "sin terminar" estén rápidamente disponibles para la comunidad científica. . La división HTGS contiene secuencias de ADN sin terminar generadas por los centros de secuenciación de alto rendimiento.
Bases de datos de proteínas
Como proteína, la globina beta está presente en bases de datos como la base de datos no redundante (nr) de GenBank (Benson et al., 2009), la base de datos SwissProt (Boeckmann et al., 2003), UniProt (UniProt Consortium 2007) y Protein Data Bank (Kouranov et al., 2006).

GNCBI: CENTRO NACIONAL DE INFORMACION DE BIOTECNOLOGIA.
El Centro Nacional de Información Biotecnológica promueve la ciencia y la salud al proporcionar acceso a información biomédica y genómica.
Misión
Comprender el lenguaje mudo pero elegante de las células vivas es la búsqueda de la biología molecular moderna. De un alfabeto de solo cuatro letras que representan las subunidades químicas del ADN, surge una sintaxis de los procesos de la vida cuya expresión más compleja es el hombre.
Creando NCBI
El fallecido senador Claude Pepper reconoció la importancia de los métodos computarizados de procesamiento de información para la realización de investigaciones biomédicas y patrocinó la legislación que estableció el Centro Nacional de Información Biotecnológica (NCBI) el 4 de noviembre de 1988, como una división de la Biblioteca Nacional de Medicina (NLM). )
Investigación
Obtiene información sobre biología molecular, la misión del NCBI es desarrollar nuevas tecnologías de la información para ayudar a comprender los procesos moleculares y genéticos fundamentales que controlan la salud y la enfermedad.
Responsabilidades
lleva a cabo investigaciones sobre problemas biomédicos fundamentales a nivel molecular utilizando métodos matemáticos y computacionales
apoya la capacitación en investigación básica y aplicada en biología computacional para becarios postdoctorales a través del Programa de Investigación Intramural NIH
desarrolla, distribuye, apoya y coordina el acceso a una variedad de bases de datos y software para las comunidades científica y médica
desarrolla y promueve estándares para bases de datos, depósito e intercambio de datos y nomenclatura biológica
Subtema
El NCBI asumió la responsabilidad de la base de datos de secuencias de ADN GenBank en octubre de 1992. El personal del NCBI con capacitación avanzada en biología molecular construye la base de datos a partir de secuencias enviadas por laboratorios individuales.
NCBI apoya y distribuye una variedad de bases de datos para las comunidades médicas y científicas. Estos incluyen la Herencia Mendeliana en Línea en el Hombre (OMIM), la Base de Datos de Modelado Molecular (MMDB) de estructuras de proteínas 3D, la Colección Única de Secuencias Genéticas Humanas (UniGene), un Mapa Genético del Genoma Humano, el Buscador de Taxonomía y el Genoma de Cáncer. Proyecto de Anatomía (CGAP), en colaboración con el Instituto Nacional del Cáncer.
Entrez es el sistema de búsqueda y recuperación NCBI que proporciona a los usuarios acceso integrado a datos de secuencia, mapeo, taxonomía y estructurales.
BLAST es un programa para la búsqueda de similitud de secuencias desarrollado en NCBI y es fundamental para identificar genes y características genéticas.
Difusión y educación
Subtema
El NCBI fomenta la comunicación científica en el área de las computadoras, según se aplica a la biología molecular y la genética, mediante el patrocinio de reuniones, talleres y series de conferencias.

Base de Datos de ADN de Japón (DDBJ)
El Centro DDBJ recopila datos de secuencias de nucleótidos como miembro de INSDC (Colaboración internacional de bases de datos de secuencias de nucleótidos) y proporciona datos de secuencias de nucleótidos y sistemas de supercomputadores disponibles de forma gratuita para apoyar las actividades de investigación en ciencias de la vida
Misión
El Centro DDBJ debe desempeñar un papel importante en la investigación en biología de la información y ejecutar la operación DDBJ en el mundo.
El propósito principal de las operaciones de DDBJ es mejorar la calidad de INSD, como dominios públicos.
Actividades principales
Construcción y Operación de INSDC
El Centro DDBJ contribuye internacionalmente como miembro de INSDC para recopilar y proporcionar datos de secuencias de nucleótidos con ENA / EBI en Europa y NCBI en los Estados Unidos. El Centro DDBJ está oficialmente certificado para recopilar secuencias de nucleótidos de investigadores y emitir el número de acceso internacionalmente reconocido para los remitentes de datos.
Proporcionar datos de secuencias de nucleótidos y aminoácidos relacionados con solicitudes de patentes.|
El INSD contiene datos de secuencia de nucleótidos relacionados con solicitudes de patentes recopiladas por las Oficinas de Patentes en Japón, Corea, Europa y Estados Unidos. El Centro DDBJ proporciona datos de secuencias de aminoácidos relacionados con las solicitudes de patentes recopiladas por las oficinas de patentes en Japón y Corea
Datos de secuencia incluidos en las solicitudes de patente.
Patente, Propiedad Intelectual y Prioridad.
Columna de patente de DDBJ
Gestión y funcionamiento del Instituto Nacional de Sistema de Supercomputación Genética.
El Sistema Nacional de Supercomputadores del Instituto de Genética (Supercomputadora NIG) es un sitio de utilización de computadoras a gran escala con el análisis del genoma como su enfoque principal.
EL sistema proporciona servicios de sistema de supercomputación que incluyen computadoras de vanguardia de tipo clúster a gran escala, computadoras de gran capacidad para compartir memoria y dispositivos de disco de alta capacidad y alta velocidad.
Brindar servicios de búsqueda y análisis de datos biológicos.
Gestión de bases de datos biológicas: herramientas para depositar y recuperar
Proporciona bases de datos mantenidas por DDBJ y otros a través de servicios web o en la Supercomputadora NIG.
Proporcionar herramientas de software para el análisis de datos biológicos.
Proporciona herramientas de software para análisis de datos desarrollados por DDBJ y otros a través de servicios web o en el Supercomputador NIG.
Curso de formación y publicación.
DDBJ organiza un curso de capacitación en bioinformática, DDBJing (en japonés), para enseñar cómo enviar datos de secuencias de nucleótidos y cómo utilizar nuestros servicios para analizar datos de ciencias de la vida.
Historia
1995 Se organizó la biblioteca de datos EMBL y se solicitó la cooperación internacional para el banco de datos de secuencia de nucleótidos a Japón.
1982 EMBL y GenBank iniciaron la cooperación internacional e invitaron a Japón a participar en su banco de datos.
1983 Con el objetivo de contribuir al banco de datos internacional para recopilar, evaluar y proporcionar datos de secuencia de nucleótidos, se inició la carga de los datos del ensayo.
1984 NIG; El Instituto Nacional de Genética se reorganizó como un Instituto de Investigación Interuniversitario.
DDBJ comenzó a trabajar en NIG.
1986 Comité Consultivo de Base de Datos de ADN organizado.
Por este lanzamiento, consideramos este año como el inicio oficial de la operación de DDBJ.
1995 Para operar DDBJ de manera más eficiente, CIB; El Centro de Biología de la Información fue establecido en NIG.
2001 CIB fue reorganizado como CIB-DDBJ; el Centro de Biología de la Información y Banco de Datos de ADN de Japón
2004 NIG fue reorganizado como miembro de ROIS; Organización de la investigación de la información y los sistemas . DDBJ también ha pertenecido a ROIS.
2005 DDBJ, EMBL-Bank y GenBank acordaron llamar a su colaboración INSDC; Colaboración internacional de bases de datos de secuencias de nucleótidos; y para llamar a la base de datos de secuencia de nucleótidos unificada INSD; La base de datos internacional de secuencias de nucleótidos.
2007 DBCLS; La base de datos del Centro de Ciencias de la Vida fue recientemente fundada en ROIS
2009 El personal docente de DDBJ ha sido enormemente reorganizado. DDBJ colabora con DBCLS más estrechamente.
2009 INSDC agregó una reunión de colaboración para tratar los enormes datos de secuencia producidos por los secuenciadores de la próxima generación (Archivo de lectura de secuencia) y los rastros producidos por los secuenciadores tradicionales (Archivo de rastreo).
2012 DDBJ, expandiendo sus actividades de banco de datos de ADN, fue reestructurado como uno de los Centros de Proyectos de Infraestructura Intelectual de NIG, al estar separado de CIB.
2013 Colaborando con NBDC; National Bioscience Database Center , DDBJ Center comenzó a operar el archivo para todos los tipos de datos fenotípicos genéticos y no identificados a nivel individual de sujetos humanos, JGA ; Genotipo japonés-fenotipo Archivo.
El Centro DDBJ está en funcionamiento en la Organización de Investigación del Sistema Nacional de Información y Sistemas de Genética (NIG) en Mishima, Japón, con el respaldo del MEXT; Ministerio de Educación, Cultura, Deportes, Ciencia y Tecnología de Japón .

CENTRO NACIONAL DE INFORMACION DE BIOTECNOLOGIA.
ESTRUCTURA
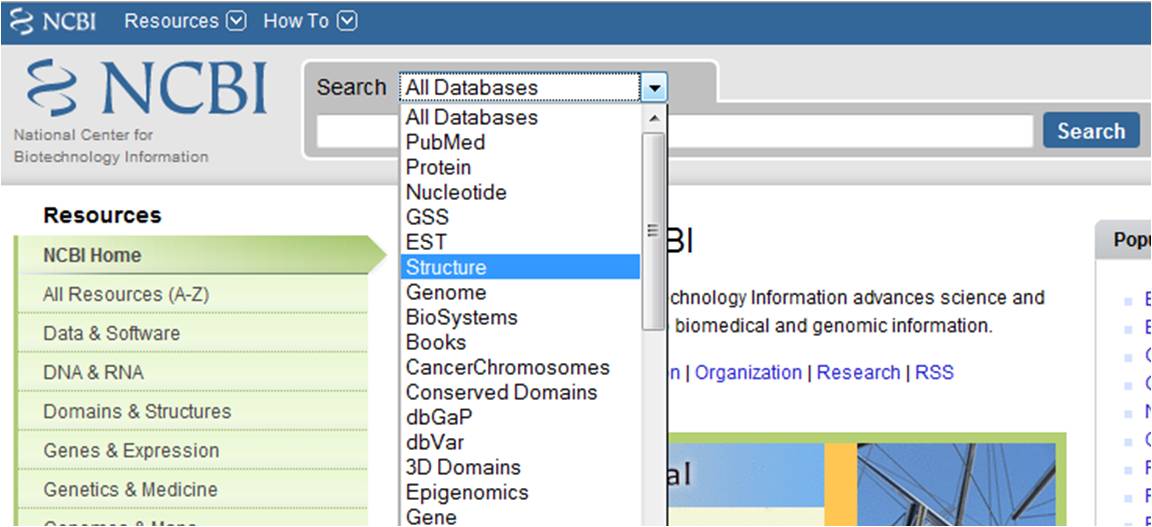
El sitio de estructura NCBI mantiene la Base de datos de modelado molecular (MMDB), una base de datos de estructuras tridimensionales macromoleculares, así como herramientas para su visualización y análisis comparativo. MMDB contiene estructuras de biopolímeros determinadas experimentalmente obtenidas del Protein Data Bank (PDB).
TAXONOMIA
El sitio web de taxonomía del NCBI incluye un navegador de taxonomía para las principales divisiones de organismos vivos (arqueas, bacterias, eucariotas y virus). El sitio presenta información de taxonomía, como códigos genéticos y recursos de taxonomía, e información adicional, como datos moleculares sobre organismos extintos y cambios recientes en los esquemas de clasificación.

LIBROS
NCBI ofrece varias docenas de libros en línea. Estos libros se pueden buscar y están vinculados a PubMed.

OMIM
La herencia mendeliana en línea en el hombre (OMIM) es un catálogo de genes humanos y trastornos genéticos. Fue creado por Victor McKusick y sus colegas y desarrollado para la World Wide Web por NCBI (Hamosh et al., 2005). La base de datos contiene información de referencia detallada. También contiene enlaces a artículos de PubMed e información de secuencias.

BLAST
BLAST (Herramienta básica de búsqueda de alineación local) es la herramienta de búsqueda de similitud de secuencias del NCBI diseñada para apoyar el análisis de bases de datos de nucleótidos y proteínas (Altschul et al., 1990, 1997). BLAST es un conjunto de programas de búsqueda de similitud diseñados para explorar todas las bases de datos de secuencias disponibles, independientemente de si la consulta es proteína o ADN.

ENTREZ
Entrez integra la literatura científica, las bases de datos de secuencias de ADN y proteínas, datos de estructura de proteínas tridimensionales, conjuntos de datos de estudios de población y ensamblajes de genomas completos en un sistema estrechamente acoplado.
ACCESO A LA INFORMACIÓN: NÚMEROS DE ADHESIÓN A LA ETIQUETA E IDENTIFICAR SECUENCIAS
Una característica esencial de los registros de secuencias de ADN y proteínas es que están marcados con números de acceso.
Un número de acceso es una cadena de aproximadamente 4 a 12 números y / o caracteres alfabéticos que están asociados con un registro de secuencia molecular.
Un número de acceso también puede marcar otras entradas, como las estructuras de proteínas o los resultados de un experimento de expresión génica.
Los números de acceso de las moléculas en diferentes bases de datos tienen formatos característicos.Estos formatos varían porque cada base de datos emplea su propio sistema.
NCBI tasigna números de identificación de secuencia únicos que se aplican a las secuencias individuales dentro de un registro.
Los números de IG se asignan consecutivamente a cada secuencia que se procesa. Por ejemplo, la secuencia de ADN de la globina beta humana asociada con el número de acceso NM_000518.4 tiene un identificador de gen GI: 28302128.
El Proyecto de Secuencia de Referencia (RefSeq)
El objetivo de RefSeq es proporcionar la mejor secuencia representativa para cada transcripción normal (es decir, no mutada) producida por un gen y para cada producto proteico normal Puede haber cientos de números de acceso de GenBank correspondientes a un gen, ya que GenBank es una base de datos de archivos que a menudo es altamente redundante.
Las entradas de RefSeq son curadas por el personal de NCBI y son casi no redundantes. Sin embargo, puede haber dos proteínas codificadas por genes distintos que compartan un 100% de identidad de aminoácidos. A cada uno se le asigna su propio identificador RefSeq único.
Las entradas de Refseq tienen diferentes niveles de estado (pronosticados, provisionales y revisados), pero en cada caso la entrada RefSeq tiene la intención de unificar los registros de secuencia.
El proyecto de secuencia de codificación de consenso (CCDS)
El proyecto Consensus Coding Sequence (CCDS) se estableció para identificar un conjunto central de secuencias codificadoras de proteínas que proporcionan una base para un conjunto estándar de anotaciones genéticas.
El proyecto CCDS es una colaboración entre cuatro grupos (EBI, NCBI, Wellcome Trust Sanger Institute y University of California, Santa Cruz [UCSC]). Actualmente CCDS se ha aplicado a los genomas humanos y de ratón, y por lo tanto su alcance es considerablemente más limitado que el de RefSeq.

COLABORACIÓN DE BASE DE DATOS DE SECUENCIAS DE NUCLEOTIDOS
La colaboración internacional de bases de datos de secuencias de nucleótidos (INSDC, por sus siglas en inglés) es una iniciativa fundamental de larga data que opera entre DDBJ , EMBL-EBI y NCBI .
El consejo asesor del INSDC, el comité asesor internacional , está formado por miembros de cada uno de los órganos asesores de las bases de datos.
Las personas que envíen datos a las bases de datos de secuencias internacionales deben conocer la política de INSDC .
ACCESO A LA INFORMACIÓN: BASE DE DATOS DE PROTEÍNICAS
Para obtener secuencias de proteínas. La base de datos de Entrez Protein en NCBI consta de regiones codificadas traducidas de GenBank, así como secuencias de bases de datos externas
SWISS-PROT, Protein Research Foundation [PRF] y el Protein Data Bank [PDB]). El EBI también proporciona información sobre proteínas a través de estas principales bases de datos.
UniProt
Subtema
Universal Protein Resource (UniProt) es el catálogo de secuencias de proteínas centralizado más completo . Formado como un esfuerzo de colaboración en 2002, consiste en una combinación de tres bases de datos clave.
Swiss-Prot considerada como la base de datos de proteínas mejor anotada, con descripciones de la estructura y función de las proteínas agregadas por expertos curadores.
La biblioteca de base de datos de secuencias de nucleótidos EMBL (TrEMBL) traducida proporciona anotaciones automáticas (en lugar de manuales) de proteínas que no se encuentran en Swiss-Prot
PIR mantiene la Base de datos de secuencias de proteínas, otra base de datos de proteínas curada por expertos.
UniProt está organizado en tres capas de base de datos.
(1) UniProt Knowledgebase (UniProtKB) es la base de datos central que se divide en UniProtKB / Swiss-Prot anotado manualmente y UniProtKB / TrEMBL anotado computacionalmente.
(2) Los clústeres de referencia UniProt (UniRef) ofrecen clústeres de referencia no redundantes basados en UniProtKB.
(3) El archivo UniProt, UniParc, consiste en un archivo estable y no redundante de secuencias de proteínas de una amplia variedad de fuentes
El sistema de recuperación de secuencias en EXPASY
El servidor ExPASy es un recurso importante para las herramientas de análisis, software y bases de datos relacionados con la proteómica. proporcionar acceso a la base de datos UniProt, ExPASy sirve como un portal para el Sistema de Recuperación de Secuencias (SRS).
Una salida consiste en un registro SwissProt. Esto proporciona información muy útil y bien organizada, incluidos nombres alternativos y números de acceso; enlaces de literatura; datos funcionales e información sobre localización celular;

Base de Datos de Secuencias de Nucleótidos del Laboratorio Europeo de Biología Molecular (EMBL)
El Laboratorio Europeo de Biología Molecular (EMBL) es una de las instituciones de investigación líderes en el mundo y el laboratorio más importante de Europa para las ciencias de la vida.
Ubicaciones EMBL
EMBL opera desde seis sitios en toda Europa:
Heidelberg, Alemania - laboratorio principal
Hinxton, Reino Unido - Instituto Europeo de Bioinformática (EMBL-EBI)
Grenoble, Francia - investigación y servicios para biología estructural
Hamburgo, Alemania - investigación y servicios para biología estructural
Roma, Italia - epigenética y neurobiología
Barcelona, España - Biología de tejidos y modelación de enfermedades.
Misiones de EMBL
realizar investigación básica en biología molecular;
capacitar a científicos, estudiantes y visitantes en todos los niveles;
ofrecer servicios vitales a los científicos en los estados miembros;
desarrollar nuevos instrumentos y métodos;
participar activamente en la transferencia de tecnología ;
e integrar la investigación europea en ciencias de la vida.
Investigación en EMBL
La investigación en EMBL enfatiza el análisis experimental en múltiples niveles de organización biológica, desde la molécula hasta el organismo, así como la biología computacional, la bioinformática y la biología de sistemas.
La investigación es realizada por más de 80 grupos independientes que cubren el espectro de la biología molecular.
Oportunidades de entrenamiento
EMBL alberga un programa completo de cursos, conferencias y talleres , muchos de ellos organizados en colaboración con la organización hermana de EMBL, EMBO (European Molecular Biology Organization).
Servicios cientificos
Los servicios proporcionados por EMBL incluyen:
Bases de datos biomoleculares y herramientas de bioinformática , en particular en EMBL-EBI;
la provisión de líneas de luz, instrumentación y tecnología de alto rendimiento para la biología estructural en las oficinas de Hamburgo y Grenoble ;
Las instalaciones básicas , que proporcionan acceso rentable y eficiente a métodos y tecnologías que son costosos de configurar o mantener, o que requieren un gasto considerable.
Ciencia y sociedad
Establecido en 1998, el objetivo principal de la iniciativa Ciencia y Sociedad en EMBL es promover una mejor comprensión de la creciente relevancia social y cultural de las ciencias de la vida.
EMBL es una organización intergubernamental especializada en investigación básica en ciencias de la vida, financiada con fondos públicos de investigación de más de 20 estados miembros , incluyendo gran parte de Europa e Israel, y dos miembros asociados, Argentina y Australia.