av Ariadna Ortega för 4 årar sedan
331
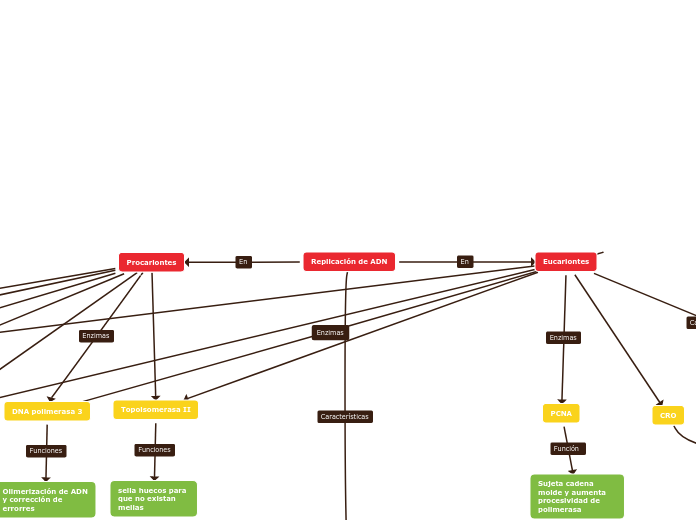
Replicación de ADN
La replicación del ADN es un proceso esencial y complejo que involucra múltiples pasos y proteínas especializadas. En organismos procariotas y eucariotas, este proceso se lleva a cabo de manera semiconservativa y bidireccional, con la síntesis de cadenas tanto continuas como discontinuas.