par Emiliano Acosta Il y a 10 mois
234
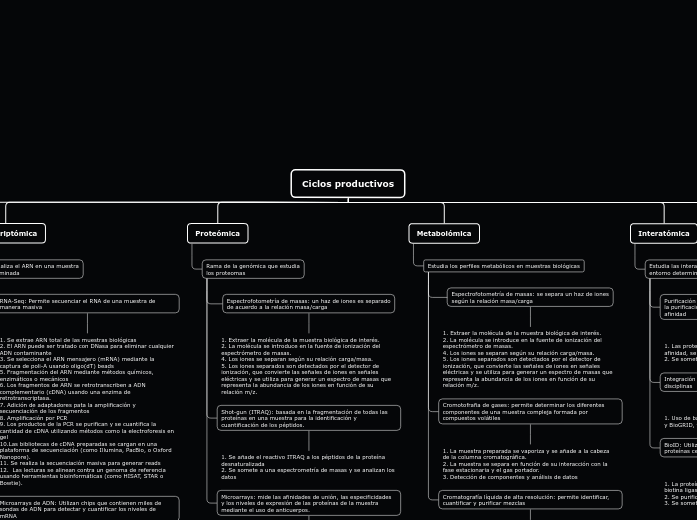
Ciclos productivos
La creciente aplicación de la secuenciación de nueva generación (NGS) ha revolucionado la genética moderna, permitiendo avances significativos en el diagnóstico y tratamiento de enfermedades.
par Emiliano Acosta Il y a 10 mois
234

Plus de détails
1. Se utiliza un chip que contiene miles de genes representados en pequeñas áreas específicas. 2. El ARNm se toma de dos muestras para comparar. 3. Se generan dos sondas marcadas diferencialmente con diferentes fluorocromos, como Cy3 y Cy5. 3. Las sondas apuntan a secuencias de genes complementarios en el chip 4. Un escáner como el Agilent G2565CA mide la intensidad de la fluorescencia
1. La proteína de interés se fusiona con una enzima como la biotina ligasa. 2. Se purifican con estreptavidina. 3. Se someten a espectrofotometría de masas.
1. Uso de bases de datos de interacción como STRING, IntAct y BioGRID, y software de análisis de redes como Cytoscape.
1. Las proteínas de interés se marcan con una etiqueta de afinidad, se purifican junto con sus proteínas asociadas 2. Se someten a una espectrofotometría de masas
1. Los componentes de la muestra se disuelven en un cartucho y se añaden al cabezal de la columna cromatográfica. 2.Los componentes de la muestra pasan a través de la columna cromatográfica gracias a la presión. 3. Se analizan los datos y la detección de componentes.
1. La muestra preparada se vaporiza y se añade a la cabeza de la columna cromatográfica. 2. La muestra se separa en función de su interacción con la fase estacionaria y el gas portador. 3. Detección de componentes y análisis de datos
1. Colocar las proteínas seleccionadas o sus anticuerpos al chip 2. Se extraen las proteínas y se marcan 3. Se añaden al chip para su complementación 4. Un escáner como el Agilent G2565CA mide la intensidad de la fluorescencia 5. Se analizan los datos
1. Se añade el reactivo ITRAQ a los péptidos de la proteína desnaturalizada 2. Se somete a una espectrometría de masas y se analizan los datos
1. Extraer la molécula de la muestra biológica de interés. 2. La molécula se introduce en la fuente de ionización del espectrómetro de masas. 4. Los iones se separan según su relación carga/masa. 5. Los iones separados son detectados por el detector de ionización, que convierte las señales de iones en señales eléctricas y se utiliza para generar un espectro de masas que representa la abundancia de los iones en función de su relación m/z.
1. Extraiga el ARN total de muestras biológicas utilizando kits de extracción comerciales y trate el ARN con ADNasa para eliminar cualquier ADN contaminante. 2. Cuantificar el ARN utilizando un espectrofotómetro 3. Evaluar la calidad del ARN mediante electroforesis en gel. 4. síntesis de ADNc 5. Reparación de mezcla de qPCR 6. Cargue la mezcla de qPCR en una placa de PCR en un ciclador térmico. 7. Después de pasar por los cicladores y medir la fluorescencia y los datos se analizan
1. Utiliza un chip que contiene miles de genes representados en pequeñas áreas específicas. 2. El ARNm se toma de dos muestras para comparar. 3. Se generan dos sondas marcadas diferencialmente con diferentes fluorocromos, como Cy3 y Cy5. 3. Se añaden sondas a secuencias de genes complementarios en el chip. 4. Un escáner como el Agilent G2565CA mide la intensidad de la fluorescencia 5. La fluorescencia en cada sonda indica la presencia y cantidad del ARNm correspondiente en la muestra.
1. Se extrae ARN total de las muestras biológicas 2. El ARN puede ser tratado con DNasa para eliminar cualquier ADN contaminante 3. Se selecciona el ARN mensajero (mRNA) mediante la captura de poli-A usando oligo(dT) beads 5. Fragmentación del ARN mediante métodos químicos, enzimáticos o mecánicos 6. Los fragmentos de ARN se retrotranscriben a ADN complementario (cDNA) usando una enzima de retrotranscriptasa. 7. Adición de adaptadores pata la amplificación y secuenciación de los fragmentos 8. Amplificación por PCR 9. Los productos de la PCR se purifican y se cuantifica la cantidad de cDNA utilizando métodos como la electroforesis en gel 10.Las bibliotecas de cDNA preparadas se cargan en una plataforma de secuenciación (como Illumina, PacBio, o Oxford Nanopore). 11. Se realiza la secuenciación masiva para generar reads 12. Las lecturas se alinean contra un genoma de referencia usando herramientas bioinformáticas (como HISAT, STAR o Bowtie).
1. Se utiliza un chip que contiene miles de genes representados en pequeñas áreas específicas. 2. Se toma el mRNA de dos muestras a comparar 3. Se generan dos sondas marcadas diferencialmente con fluorocromos diferentes, como Cy3 y Cy5 3. Las sondas se a las secuencias de genes complementarios en el chip 4. Un escaner como el Agilent G2565CA mide la intensidad de la fluorescencia
1. Corroborar la calidad de los datos 2. Se identifican todos los pares de reads que se solapan lo suficientemente bien 3. Se calculan los solapamientos entre los reads para determinar cuáles tienen secuencias iguales 4. Construcción del gráfico de solapamientos llamado Layout 5. Se utilizan algoritmos de ensamblaje que aprovechan la estructura del gráfico que corresponden a segmentos del genoma que están siendo ensamblados 6. Se verifica la calidad del ensamblaje y se realizan ajustes si es necesario.
Una read es una secuencia corta de nucleótidos que se generan en una secuenciación de ADN
1. Segmentación de ADN 2. Marcaje del ADN por medio de primers 3. Amplificación de fragmentos de ADN mediante PCR 4. Secuenciación de los fragmentos 5. Reconstrucción de la secuencia completa por medio de secuencias de referencia y exportación a ficheros de almacenamiento de datos