によって Izadora Oliveira Soares 4年前.
262
Organigrama
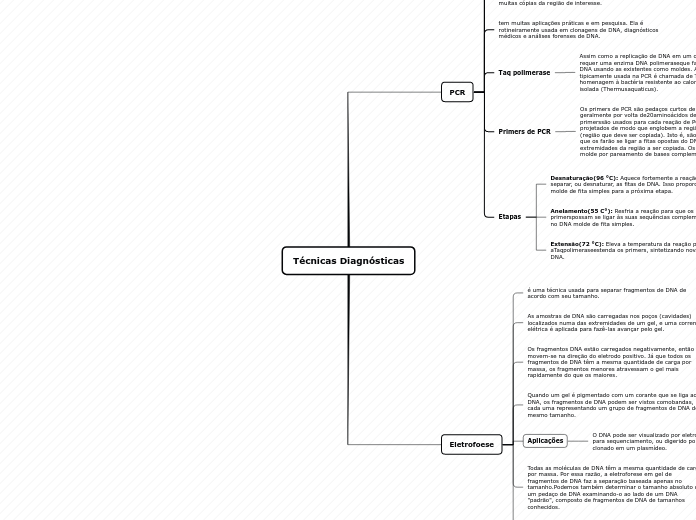
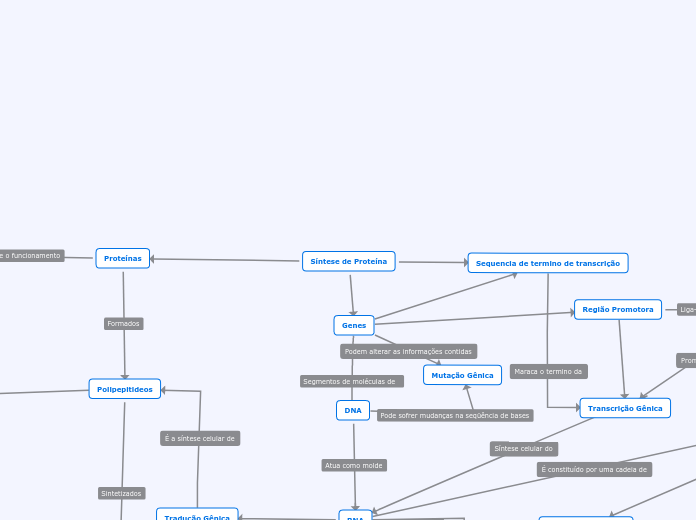
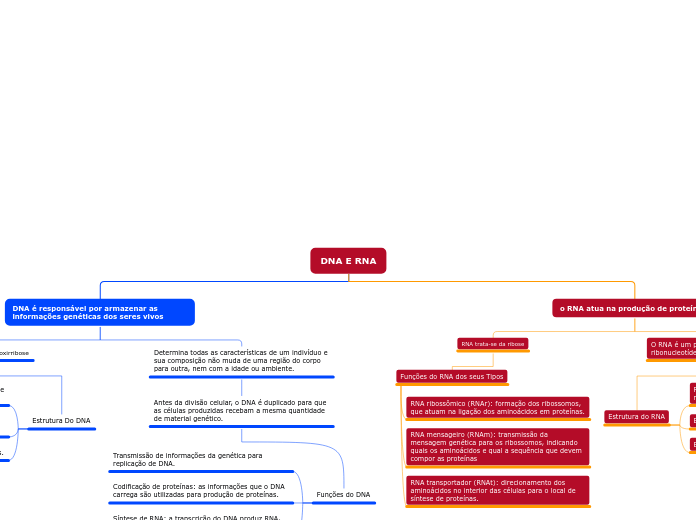
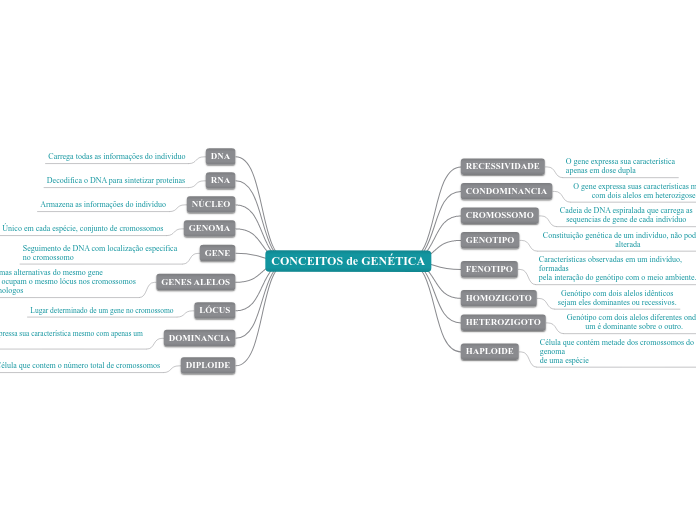
A técnica de PCR é utilizada para amplificar regiões específicas do DNA em laboratório. Para isso, são usados primers, que são pedaços curtos de DNA de fita simples que se ligam a sequências complementares no DNA molde.