Luciferasa
Tecnologías de secuenciación
NGS: Secuenciación de nueva generación.
Aplicaciones.
Metagenómica para estudiar ecología microbiana.
Analizar cambios en los mecanismos reguladores del genoma.
Analizar cambios epigenómicos.
Analizar resultados de transcriptómica
Medición de la variación genética de un organismo que ya se conoce,
Alinear secuencias a secuencias que no se conozcan en "Sequencing de novo"
Plataformas de secuenciación.
Illumina.
Características.
Son raros los errores por inserción o deleción.
El error de sustitución es el error más común.
Es frecuente en secuencias con alto contenido de GC.
La tasa no es uniforme y puede incrementar.
Los reads son de una longitud fija.
Resultados.
4 fotos x ciclo del chip
Proceso de secuenciación.
Extiende la cadena con
"Chips" de vidrio que poseen 8 canales.
Tiene adherido primers.
Hibridación con una molécula
Separación y eliminación de la cadena original.
Extensión de la cadena naciente o complementaria.
Forma un puente o "bridge" a un primer adyacente.
Se deshacen y se inhibe la secuenciación por
Se bloquean los extremos 3'
Emite luz por láser.
Cambios de temperatura.
Se une la polimerasa sintetizando una nueva cadena
Se repite el anterior proceso exponencialmente.
Un Cluster.
Subtopic
"Real Time Sequencing: Oxford Nanopore Technologies."
El gradiente de corriente
Nucleótidos llamados "K-meros"
"SMRT single Molecule Real Time."
Hay dos tipos:
"Long Reads."
"Pacific Biosciences"
Usa 4 fluoróforos unidos a nucleótidos.
El grupo fosfato es cortado
Fluorescencia.
"Short Reads."
Ensable de fragmentos.
Silico.
"Cyclic reversible termination."
Nucleótidos de terminación virtuales.
Nucleósido modificado
No estar unido a nucleótido de cadena naciente.
Elongación de la cadena.
Estar unido a nucleótido de cadena naciente.
Inhibición de elongación de la cadena.
Incorporación de nucleótidos marcados.
Se repite
La eliminación e incorporación sucesiva
Templates unidos a adaptadores y primers universales.
Inicia la inserción de nucleótidos (cada un con diferente color)
Se realiza lavado.
Obtención de nucleótidos que se incorporan a la cadena naciente.
Utiliza bases del Método de Sanger.
"Sequencing by ligation."
Se repite x 10 ciclos
La eliminación e incoporporación sucesiva
Fluoróforos.
Secuenciamiento de templates.
Ligasas.
Liga la sonda (marcada con fluoróforo) con el template y primer universal.
Alineamiento.
"Ion torrent technology."
Se puede hacer de dos formas:
Adición de T'S en los pocillos.
Coindicen con la secuencia original.
Dos iones de hidrógeno.
Cambio mayor en el pH de la dilución
Traducido a Base-cooling.
Emulsión PCR.
Placa picotituladora que poseen microperlas.
Se le añaden nucleótidos a la cadena naciente.
Iones de hidrógeno.
Cambios en el pH de la dilución
Detectado por un micropontenciómetro.
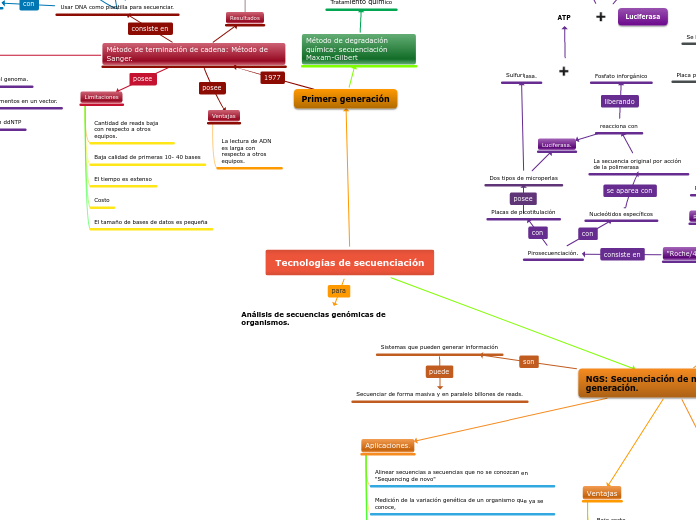
"Roche/454 Sequencing."
Pirograma.
Pirosecuenciación.
Nucleótidos específicos
La secuencia original por acción de la polimerasa
reacciona con
Fosfato inforgánico
Placas de picotitulación
Dos tipos de microperlas
Sulfurilasa.
Luciferasa.
Preparación.
Amplificación por PCR
Emulsion PCR.
Emulsión en un tubo con
Oligonucleótidos
Polimerasa
"Bridge PCR"
Unión de un primer a una secuencia de interés.
Unión de adaptadores.
Formación de librerías.
El proceso es más rápido.
No se necesita realizar electroforesis
Eficiente
Las lecturas son más largas.
Bajo costo
Sistemas que pueden generar información
Secuenciar de forma masiva y en paralelo billones de reads.
Primera generación
Método de degradación química: secuenciación Maxam-Gilbert
Tratamiento químico
Roturas en 1 o 2 bases de nucleótidos.
Cuatro reacciones.
Método de terminación de cadena: Método de Sanger.
Preparación
Secuenciación con ddNTP
Clonación de fragmentos en un vector.
Fragmentación del genoma.
Ventajas
La lectura de ADN es larga con respecto a otros equipos.
Resultados
Cromatograma
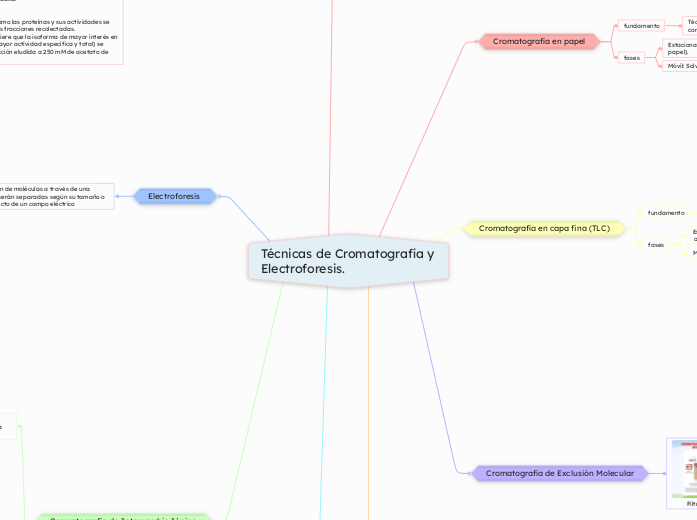
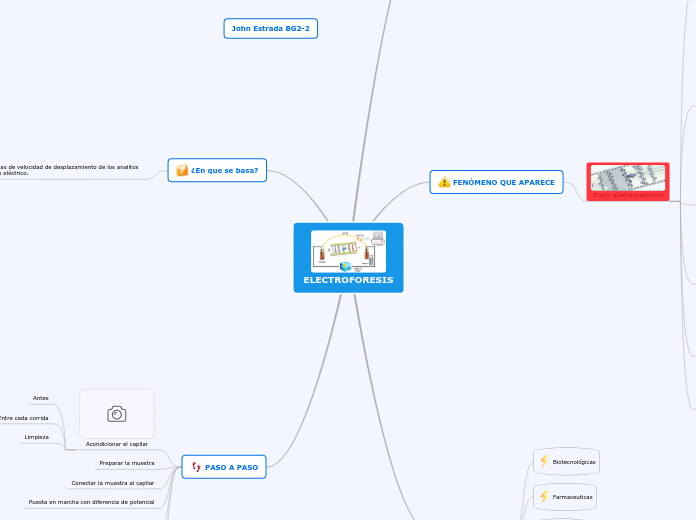
Electroforesis
Limitaciones
El tamaño de bases de datos es pequeña
Costo
El tiempo es extenso
Baja calidad de primeras 10- 40 bases
Cantidad de reads baja con respecto a otros equipos.
Usar DNA como plantilla para secuenciar.
DNA polimerasa
Incorpora dNTP a la nueva cadena.
ddNTP (modificado)
Fluoróforo
Da un color determinando la terminación de la cadena.
Láser en un capilar.
Electroforesis capilar
Migración de moléculas de una muestra de acuerdo a su tamaño
Posee un H
Terminación de la elongación de la nueva cadena.
dNTP
Posee un grupo OH
La unión a otro dNTP para la elongación de la nueva cadena.
Primer
Permite la elongación de la nueva cadena.
Análisis de secuencias genómicas de organismos.