Hecho por: Brenda Viridiana González Moya
Génetica clínica
Grupo: 3722
Referencias: Salazar Montes, A. M., Sandoval Rodríguez, A. S., & Armendáriz Borunda, J. S. (2016).
Biología Molecular. Fundamentos y aplicaciones en las ciencias de la salud, 2e (3.a ed.). McGRAW-HILL/INTERAMERICANA EDITORES, S.A. DE C.V.
Aula Virtual de biología. (s.f). Aula Virtual de Biología. UM.es. https://www.um.es/molecula/dupli.htm
Traducción
Una vez termianda la traducción ocurre la topogénesis
la ruta que siguen las proteínas en la célula hasta alcanzar su localización (intracelular o extracelular) donde ejercerán su función. Esta topogénesis se realiza en el citosol.
La clave del proceso es la péptido señal
modificaciones postraduccionales
La expresión de un gen termina hasta la generación de una proteína madura y activa
Acetilación
Metilación
Fosforilación
Sulfatación
Glicosilación
Hidroxilación
Acilación
Prenilación
Formación de puentes disulfuro
Procesamiento proteolítico
componentes del complejo traduccional
rRNA
más abundante en las células y forma parte de los ribosomas
los ribosomas
Estructura
Sitios A y P:participan de forma directa en la decodificación del mRNA
El P se sitúa el peptidil-tRNA se lleva a cabo la elongación de la cadena peptídica, mientras que el sitio A es el lugar por el cual el aminoacil-tRNA (correspondiente a la lectura del codón) llega al complejo traduccional, para después transferir este aminoácido al peptidil-tRNA y generar el enlace peptídico.
Sitio E ugar por el cual saldrá del complejo ribosomal el tRNA sin aminoácido, una vez que lo dejó en la cadena polipeptídica en formación.
Cataliza la transferencia del aminoacil-tRNA al peptidil-tRNA , siguiendo la secuencia de codones del mRNA, según las equivalencias del código genético.
la unión del mRNA al tRNA, proporcionando los sitios en los que interactúa el codón del mRNA con el anticodón del tRNA.
tRNA
pequeños RNA con estructura de bucles y son los encargados de transportar al aminoácido hasta el ribosoma
en su secuencia al anticodón
mRNA
información genética almacenada en el DNA y su secuencia es complementaria a una de las cadenas del DNA
cadena molde que la traducción requiere para su inicio es el mRNA
se codifica por el código genético
código génetico
Código genético degenerado y casi universal
muestra de manera práctica los 64 codones y su significado
61 son utilizados para codificar a los 20 diferentes aminoácidos
Los 3 restantes son usados terminan la traducción (UAA, UGA, UAG)
Utilizado por la célula en que la secuencia de nucleótidos de mRNA se convierte de manera secuencial y sin interrupciones en una secuencia de aminoácidos
se codifica la información es en triplete o codón, que representará a un aminoácido o señal específica
síntesis de una proteína de acuerdo con la información genética y se emplea una molécula de mRNA como molde
Se interpreta la información contenida en el mRNA a través de la decodificación
lectura del mRNAtiene como inicio el extremo 5′ y finaliza en el extremo 3
traducción
terminación
Una vez finalizada la síntesis de una cadena polipeptídica, el mRNA queda libre y puede leerse de nuevo
varios ribosomas leen una misma molécula de mRNA de forma simultánea
Poliribosoma o polisoma
El sitio A se encuentra en alguno de los codones de paro del mRNA, los cuales tienen como característica no codificar ningún aminoácido.
los factores de liberación o de terminación (RF) imitan al tRNA y reconocen directamente el codón de terminación
RF requieren de una molécula de GTP para permitir que el polipéptido recién sintetizado se libere del complejo traduccional y, al mismo tiempo, permite que se disocie la unión entre el rRNA y el mRNA
elongación
implica la incorporación de nuevos aminoácidos mediante la formación de enlaces péptidicos
Reacción ciclíca
c. Desplazamiento del ribosoma
b. Transferencia del aminoácido al peptidil-tRNA
a. Decodificación del aminoacil-tRNA en el sitio A
2 factores de elongación que están unidos al GTP
EF-Tu (en bacterias) y EF-1a (en eucariotas), asociada al aminoacil-tRNA y con un GTP para formar un complejo ternario
catalizados por la enzima peptidiltransferasa que se encuentra en la subunidad mayor del ribosoma
La iniciación termina cuando el complejo ribosomal está completo y está formada la unión codón/anticodón
unión de la subunidad menor al mRNA con la asistencia de factores de iniciación (IF)
Una vez unido el ribosoma al mRNA a través del rRNA, el tRNA iniciador entra al sitio P y reconoce al codón AUG para iniciar la traducción
La identificación del sitio P es mediada por la acción de los IF; sólo bajo estas condiciones el tRNA iniciador es el único tRNA que puede entrar por el sitio P; los siguientes entrarán por el sitio A.
La lectura del codón se lee en dirección 5′ a 3′ por el anticodón
En el acople de la subunidad menor del ribosoma con el metionil-tRNA actúa el eIF-4 para eucariotas y el IF-3 para procariotas
IF-2 se asocia con GTP y se une al metionil-tRNA con el complejo ribosomal; si el codón/anticodón son complementarios, se hidroliza el GTP y la unión se vuelve estable.
En procariontes se encuentra la secuencia de Shine-Dalgarno y en eucariotas la secuencia de Kozak
reconocimiento del codón de inicio
procesos para la formación del complejo traduccional y la formación del primer enlace peptídico
Biosíntesis de proteínas (inicio): El mRNA se une a la subunidad menor. A éstos se asocia el aminoacil-tRNA,Después se une la subunidad ribosómica mayor, formándose el complejo ribosomal.
El primer codón que se traduce es generalmente el AUG, que corresponde con el aminoácido metionina en eucariotas y formilmetionina en procariotas.
activación de los aminoácidos
Activación de los aminoacil-tRNA. Proceso que requiere de la hidrólisis de ATP en dos reacciones secuenciales, que se catalizan en la enzima, la aminoacil-tRNA sintetasa. Primero, la enzima une el aminoácido al fosfato del ATP con la liberación concomitante de pirofosfato. Esto se llama un intermediario aminoacil-adenilato. En el segundo paso, la enzima cataliza la transferencia del aminoácido a los OH (2′ o 3′) de la porción de ribosa del residuo de adenosina 3′ terminal del tRNA generando un aminoacil-tRNA activado. Aunque esta reacción es libremente reversible, la reacción hacia delante es favorecida por la hidrólisis acoplada del PPi.
acción de la enzima aminoacil-tRNA-sintetasa y la hidrólisis de dos moléculas de ATP , uno para la remoción del PPi y el segundo para la hidrólisis de PPi a 2Pi con la intervención de la enzima pirofosfatasa
que un aminoácido pueda unirse a su tRNA específico y genere un aminoacil-tRNA cargado
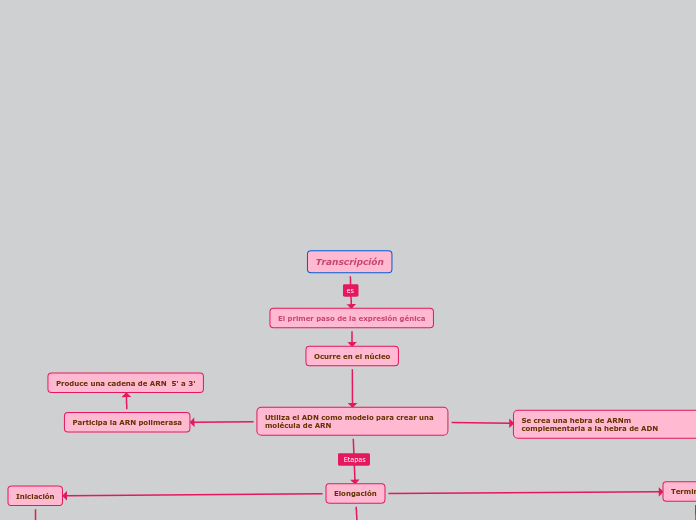
Transcripción
El desarrollo y el fenotipo de un organismo pueden regularse por el producto génico que interactúa con otros genes o con el ambiente en tiempo y espacio
Procesamiento de RNA
Para la maduración del
transcrito primario
Incluye
Proceso de edición
forma de modificación postranscripcional del mRNA y se presenta en ciertos genes, tejidos y tipos celulares
Mecanismos
La inserción o deleción de una uridina dirigida por un RNA guía se ha observado en procariotas y ha cambiado los marcos de lectura para la traducción.
cambio de base adenina por inosina, que prefiere aparearse con citosina (enzima adenosina desaminasa de acción sobre RNA, ADAR)
esaminación oxidativa de una citosina metilada, que se convierte en uridina del codón CAA (enzima citidina desaminasa) y genera el codón de paro UAA
adición de un capuchón de guanina modificada en el extremo 5′
A: adición de la caperuza o casquete en el extremo 5′. La base modificada 7 metilguanosina (7mG) se añade en 2′-OH de la última base del RNA, eliminando un grupo fosfato con la formación de un enlace entre carbono 5′-5′ de las ribosas.
B: modificación del extremo 3′ con la adición de la cola de Poli-A en presencia de los factores CPSF y CstF, al reconocer la secuencia AAUAAA presente en el mRNA.
C: eliminación de intrones y empalme de exones (splicing), con la formación del ayustosoma con ataque nucleofílico y transesterificación.
corte y empalme
(splaning)
Remoción de intrones y
empalme de los exones
Las regiones en el hnRNA reconocidas por el espliceosoma son secuencias conservadas de nucleótidos específicas que indican el inicio y terminación de un exón/intrón
poliadenilación del extremo 3′
implica el reconocimiento de una secuencia hexamérica AAUAAA o secuencia similar
se leería como la señal de poliadenilación que determina el final de la adición de nucleótidos a la cadena y la desintegración del complejo de transcripción
TFIIE y TFIIH, que se unen corriente arriba de la RNA pol II
Una vez unida TFIIE, se pueden unir TFIIH, que de forma excepcional continúa unido a la RNA pol II
Los nucleótidos se añaden de forma covalente al extremo 3′OH y en la región desenrollada se forma un híbrido DNA-RNA
La fosforilación del dominio carboxiterminal (CTD) está implicado en el abandono del promotor basal y el inicio de la fase de elongación
ATPasa, helicasa y cinasa, que puede fosforilar el dominio CTD de la RNA pol II
El inicio termina cuando la enzima comienza a alargar la cadena y abandona el promotor con el complejo de iniciación
La fase de inicio puede retrasarse por la ocurrencia de intentos abortivos
La RNA pol II permanece en el promotor mientras sintetiza los primeros nueve enlaces
Síntesis de los primeros enlaces nucleotídicos de RNA
Los Factores transcripcionales (AF)
Inducibles
requerido para la expresión de un determinado gen es particular para cada promotor con función reguladora
Basales
requeridos para el inicio de la transcripción en todos los promotores basales
El reconocimiento del promotor basal
inicia con la unión de la primera proteína del complejo del TFIID a la caja TATA, conocida como proteína de unión específica de TATA (TBP)
en el posicionamiento de la RNA pol II y delimita la distancia desde el punto de inicio hasta la caja TATA.
TFIID que está formado por 11 TAF
TFIIA controla la capacidad de unión de TBP al DNA y permite al TFIID reconocer la región que se extiende hacia el extremo 5′
TFIIB se une de forma adyacente a TBP y proporciona mayor superficie de reconocimiento para el anclaje de la RNA pol II
TFIIF es el medio de unión de la RNA pol II al complejo de transcripción.
La proteína TFIIH tiene actividad helicasa y contacta con la RNA pol II, lo que le permite su anclaje a ésta.
Los TAF son subunidades diferentes y pueden reconocer una variedad de promotores tanto basales como distales.
provoca una deformación en la estructura del DNA sin separar las dos cadenas
promotores que carecen de caja TATA, la TBP puede incorporarse por asociación a otras proteínas que reconocen el DNA
En el sitio de inicio de la transcripción, el DNA se separa de forma transitoria en dos cadenas sencillas; una se utiliza como molde para la síntesis de RNA.
Conforme la RNA pol avanza y copia el DNA, éste regresa a su forma de doble hélice
Tiene lugar en el núcleo
En Eucariotas para la maduraciónexiste un sistema de nucleasas (FEN1/RTH1 + RNasa H1) encargado de eliminar el cebador de RNA.
la DNA pol δ o DNA pol ε, elonga el extremo 3′ del fragmento adyacente y rellena el lugar que antes ocupaba el cebador hasta alcanzar el extremo 5′ del fragmento contiguo
La DNA ligasa I sella la mella resultante al unir el 3′-P del primer fragmento con el 5′-P del segundo
Una vez sintetizado el DNA, éste se unirá con las proteínas histonas y no histonas necesarias para la compactación de las cadenas de DNA
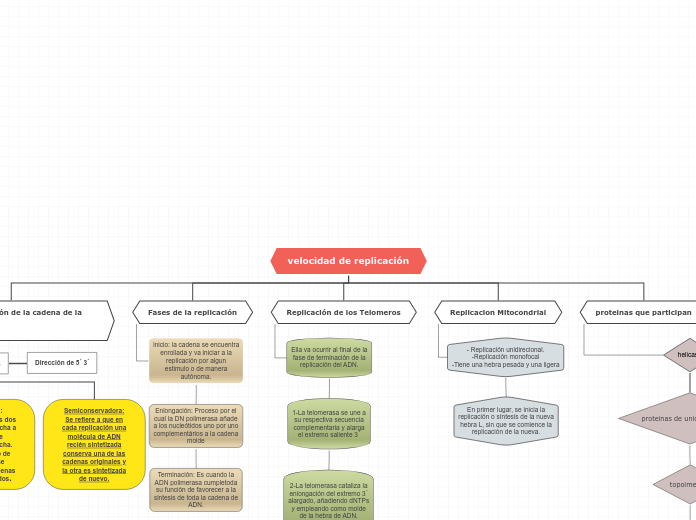
Replicación
Fases
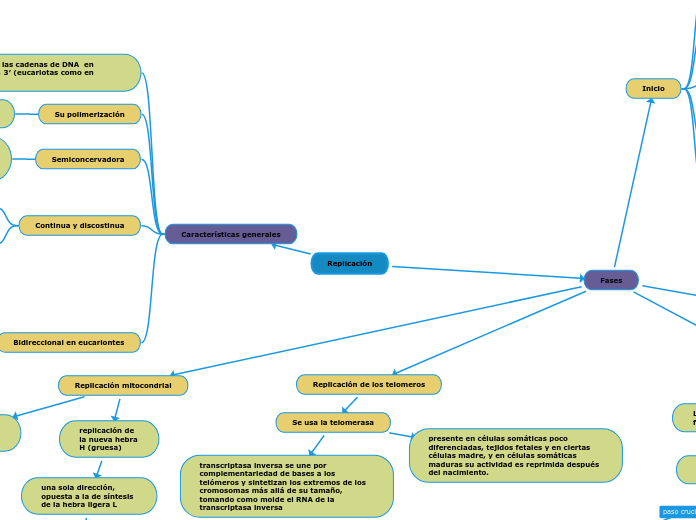
Replicación mitocondrial
replicación de la nueva hebra H (gruesa)
una sola dirección, opuesta a la de síntesis de la hebra ligera L
Su síntesis inicia cuando se han sintetizado alrededor de dos tercios de la nueva hebra L
se inicia la replicación o síntesis de la nueva hebra L (ligera)
unidireccional y avanza desplazando a la otra hebra
Replicación de los telomeros
Se usa la telomerasa
presente en células somáticas poco diferenciadas, tejidos fetales y en ciertas células madre, y en células somáticas maduras su actividad es reprimida después del nacimiento.
transcriptasa inversa se une por complementariedad de bases a los telómeros y sintetizan los extremos de los cromosomas más allá de su tamaño, tomando como molde el RNA de la transcriptasa inversa
Mecanismo de acción
se une a una secuencia complementaria en el telómero y alarga el extremo saliente 3′; posteriormente se produce un apareamiento de bases entre el DNA telomérico y el RNA de la telomerasa.
cataliza de nuevo la elongación del extremo 3′ alargado, al añadir dNTP y emplear como molde la hebra de RNA de la propia telomerasa.
la telomerasa se desliza sobre el extremo 3′ previamente elongado, conservando la complementariedad gracias a la translocación
La telomerasa elonga nuevamente el extremo 3′ saliente y se repiten los pasos de translocación y elongación
Segunda fase, se sintetiza un cebador en la hebra 5′ complementaria al telómero; la DNA polimerasa δ rellena la hebra y la ligasa sella la mella que queda en el segmento elongado del telómero bicatenario.
elimina el último cebador
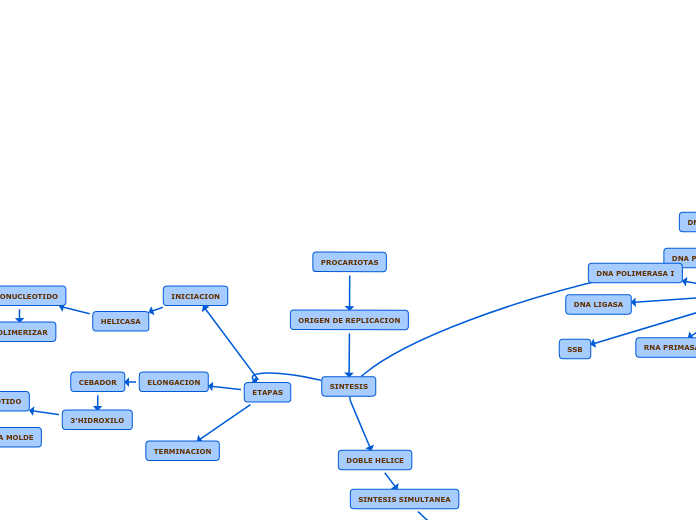
Inicio
En procariotas el primosoma crea el cebador de RNA
La primasa sintetiza un cebador y la DNA polimerasa incorporará los nucleótidos en forma complementaria a las bases de la cadena molde
Cada burbuja de replicación posee dos horquillas de replicación y la helicasa se unirá a la cadena de DNA e hidrolizará los puentes de H y por la unión de proteínas estabilizadoras RPA en eucariotas y SSB en procariotas.
sitios en que ha de comenzar son reconocidos por proteínas llamadas proteínas de reconocimiento del sitio de origen. En eucariotas, los orígenes de replicación se llaman secuencias de replicación autónoma.
Zonas en el DNA en las que se producen las burbujas de replicación no son aleatorias
Elongación
Para la síntesis de cada fragmento de Okazaki se requiere de un cebador intercalado entre estos fragmentos, mientras que en la cadena continua es necesaria la presencia de un solo cebador.
Una vez presente la maquinaria de inicio de la replicación, la horquilla avanza
Para evitar la tensión intervienen las topoisomerasas (I y II) que cortarán los enlaces fosfodiéster de la doble hélice y volverán a unirla
La DNA polimerasa añade uno por uno nucleótidos complementarios a la cadena molde
Ayudada por la PCNA que la mantiene en contacto con la cadena molde
Terminación
La DNA polimerasa δ llega al extremo del fragmento de DNA.
desacoplamiento de todo el replisoma y la finalización de la replicación
Eliminación de los cebadores, elongación del fragmento de DNA adyacente y unión de los extremos resultantes para formar una cadena continua
completar la síntesis de la cadena retardada y unir los fragmentos de Okazaki para su maduración
Características generales
Bidireccional en eucariontes
del sitio de origen de la replicación (ORI) se sintetizan las dos cadenas en ambos sentidos
Sitios ORI
Replicación monofocal
Replicación del DNA circular
Replicación multifocal
DNA mitocondrial
ORI-H y ORI-L para la cadena pesada y ligera del DNA de forma respectiva
secuencias específicas ricas en A y T que controlan la replicación del replicon
puntos de crecimiento (horquillas de replicación)
Continua y discostinua
Hebra retardada (discontinua)
se sintetiza en sentido contrario al avance de la horquilla y en fragmentos de Okazaki
Hebra adelantada (continua)
cadena que se sintetiza en el sentido que avanza la horquilla de replicación
Semiconcervadora
Cada replicación de una molécula de DNA recién sintetizada conserva una de las cadenas originales; la otra se sintetiza de novo.
Su polimerización
unión de un dNTP complementario a la hebra molde
La síntesis de las cadenas de DNA en dirección 5′ → 3′ (eucariotas como en procariotas)