Splicing alternativo: taglia esoni e mantiene introni (97% geni coding)
si può modificare anche le sequenze 5'-3' UTR
si influenza la stabilità, la posizione e l'efficienza del mRNA
ex. geni: SLO (da diversa sensibilità ai suoni nella coclea), DSCAM (38k proteine), Calcitonina (2 proteine diverse a seconda del tessuto)
spiega discrepanza nr. geni V.S. proteine
1/3 inserisce un cordone di stop prematuro creando proteine no senso che vengono degradate (grazie a meccanismo non-sense medieted mRNA decay)
controllo quantitativo dell'espressione genica
80% su sequenza codificante, 20% su UTR
10% influenza direttamente domini funzionali delle proteine (eliminandoli/inserendoli)
90% delle modifiche su CDS sembra influenzino la struttura proteica creando anse fondamentali per l'interazione con altre proteine
controllo interattoma
promotori alternativi: primo esone con più isoforme perché con diverso 5' UTR, possono essere interni e creare proteine tronche, scelti in base allo status funzionale (ex. distrofina)
scelto in base struttura promotore, elementi in cis e modifiche epigenetiche
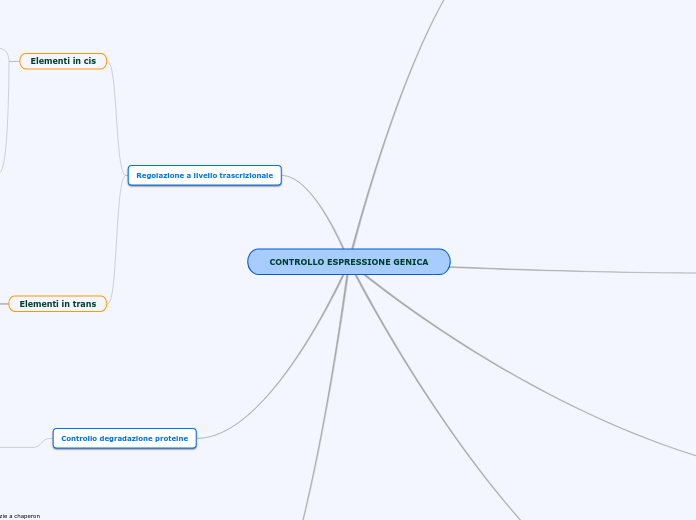
CONTROLLO ESPRESSIONE GENICA
Controllo e regolazione attività proteica
controllo quantità di proteine prodotte
autoregolazione della protezione
confinando gli enzimi in compartimenti
regolando velocità di degradazione
regolando espressione del gene che lo codifica
assemblaggio in complessi
macchine proteiche, condensati intracellulari
inibizione/attivazione retroattiva
legando enzimi chiave
fosforilazione (ATP-GTP)
modifica covalente tramite chinasi e fosfatasi, cambia fortemente la forma, reversibile (ex gtp con gef)
trasporto in situ
proteolisi
glicosilazione
ripiegamento: grazie a chaperon
Controllo degradazione proteine
la cellula controlla la longevità proteica grazie a ubiquitinizzazione e a proteosomi
il proteosoma ha un opercolo per legare ubiquitina e srotolare proteina, un cilindro che scinde la proteina (grazie a proteasi nelle pareti) e un altro opercolo in cui esce
Regolazione a livello trascrizionale
Elementi in trans
famiglie di proteine :
hanno sito legami x DNA e per le altre proteine, forme:
helix-loop/turn-helix
a dito di zinco
cerniere di leucine
la selezione delle proteine regolatrici determina il tipo di risposta cellulare
effetto combinatorio sullo stesso gene
effetto coordinatorio su più geni
fattori di trascrizione specifici
a questi si legano coattivatori, acetilano DNA o incrementano attività RNA Pol, e corepressori, deacetilano o metilano DNA
legano intensificatori e silenziatori a valle e a monte del gene
si legano al complesso di inizio grazie al mediatore che avvicina le proteine incurvando la sequenza, lega anche acetilasi per istoni e complesso di rimodernamento cromatina
fattori trascrizione basali
sempre coinvolti, si legano in maniera sequenziale
TFII D (comprende TBP), TFII A-B, RNA Pol II, TFII H (stacca RNA Pol e inizia la trascrizione, elicasica/chinasica)
Elementi in cis
sequenza trascrivibile
sequenze regolative
elementi di risposta
ex. cAMP,HRE: questo è interno al promotore, il legame al complesso recettore-ormone steroideo recluta coattivatori della RNA polimerasi II
isolatori
silenziatori
lontani ma anche in mezzo a introni
intensificatori
lontani
promotore: indica filamento, lega complesso pre-inizio, regola RNA polimerasi
Sottoargomento
DPE
non con TATA
INR
insieme a TATA
BRE
riconosce TFIIB
isole CpG
nei geni housekeeping, inattivano trascrizione se metilate
TATA box
lega TBP => induce curvatura elica per fattori di trascrizione
Controllo a cavallo tra stabilità mRNA e traduzione
Ex. = sistema ferretina-aconitasi
-ferretina sequestra ferro libero nel sangue -------transferrina porta ferro nelle cellule -aconitasi lega ferro e si lega a elemento di riposta del ferro sul gene per la transferrina e ferretina
se [Fe] aumenta, la transferrina lega aconitisa inibendola
se [Fe] cala, aconitasi lega 5' UTR della ferretina e ne blocca la traduzione, lega anche 3' UTR x gene transferrina rendendola + stabile e traducendola +
Controllo su degradazione messaggero
emivita messaggero dipende da 3'UTR, se contiene tante AU = degradato + velocemente viceversa se ha tanti C, e poli A
meccanismi di degradazione
taglio eneolitico
deCapping
POLI A deamilasi
Controllo sul trasporto e locazione
come?
degradazione generale con protezione locale
trasporto diretto usando citoscheletro
trasporto casuale e concentrazione locale
locazione grazie a sequenze lette durante la traduzione
serve per:
crea [proteine] localizzate x polarità cell.
crea domini citoplasmatici /=
sviluppo asimmetrico feto
sequenza ZIP code nel 3' UTR che invia a compartimenti endocellulari specifici
sequenza all'n terminale che porta la proteina con i ribosomi sul RER o nel citosol contestualmente alla sintesi
passaggio tra nucleo e esterno attraverso i pori nucleari = leggono la sequenza e decidono se farla passare o meno
si usano esportine (carioferine) che cooperano con Ran-GTP e NXF
grazie ciclo di traduzione primario mRNA maturo, decadimento mediato da codoni non senso
se si trova un cordone di stop prematuro cioè prima di un complesso di giunzione esone-esone, dovuto ad un introne non tagliato, si degrada il messaggero
Controllo maturazione mRNA
avvengono contemporaneamente alla trascrizione
modifiche 5' e 3' del mRNA
poli A 3'
aiuta passaggio al citoplasma, stabilità mRNA
CAP 5' = 7-metil-guanosina,
impedisce degradazione, segnale per aggancio ribosoma e per efficienza traduzione
Splicing
eccezioni allo splicing geni di:
mitocondri, tRNA, IFN, istoni, recettori ...
si regola controllando velocità trascrizione, regolando proteine per splicing sul filamento/istoni
esoni forti e deboli: i deboli non vengono integrati se la trascrizione è veloce, se la cromatina è compatta la trascrizione è lenta => gli esoni deboli vengono messi
sequenze enhancer e silencer dello splicing possono essere sia introniche che esotiche
ESS,ISS (silencer)
legano hnRNP, impediscono legame dei fattori di splicing, compattano introni
ESE,ISE (enhancer)
lega proteine SR (ricche di Serena e Arginina) che richiamano U1-2 e snRNA al confine esone-introne
si legano sequenze introniche: AG all'inizio e GU alla fine,si forma laccio (grazie ad "A" nella sequenza di rimozione dell'introne) e poi taglio tramite transesterificazione
si mette complesso di giunzione dell'esone
grazie a spliceosoma formato da snRNA + proteine (U 1-2-5-6), la maggior parte del sito catalitico è formato da RNA
perché esistono gli introni (intron late: disaccoppiano trascrizione da traduzione)
favorire la ricombinazione esonica per creare nuovi geni (exon shuffling)
splicing alternativo
contengono sequenze regolative (ISE/ISS)
danno RNA non codificanti (snoRNA, miRNA)